Quick Start Guide for HDF5 in python
HDF(Hierarchical Data Format)는 대용량 데이터를 저장하고 구성하기 위해 고안된 일련의 파일 형식 (HDF4, HDF5)입니다. 빅데이터 분석의 중요성이 부각되는 요즘, HDF는 점점더 인기있는 파일 형식이 되고 있습니다. 이 글은 파이썬에서 HDF5의 사용방법을 설명하는 간단한 예제입니다. 영문으로 된 원문은 여기를 보시기 바랍니다.
Core concepts
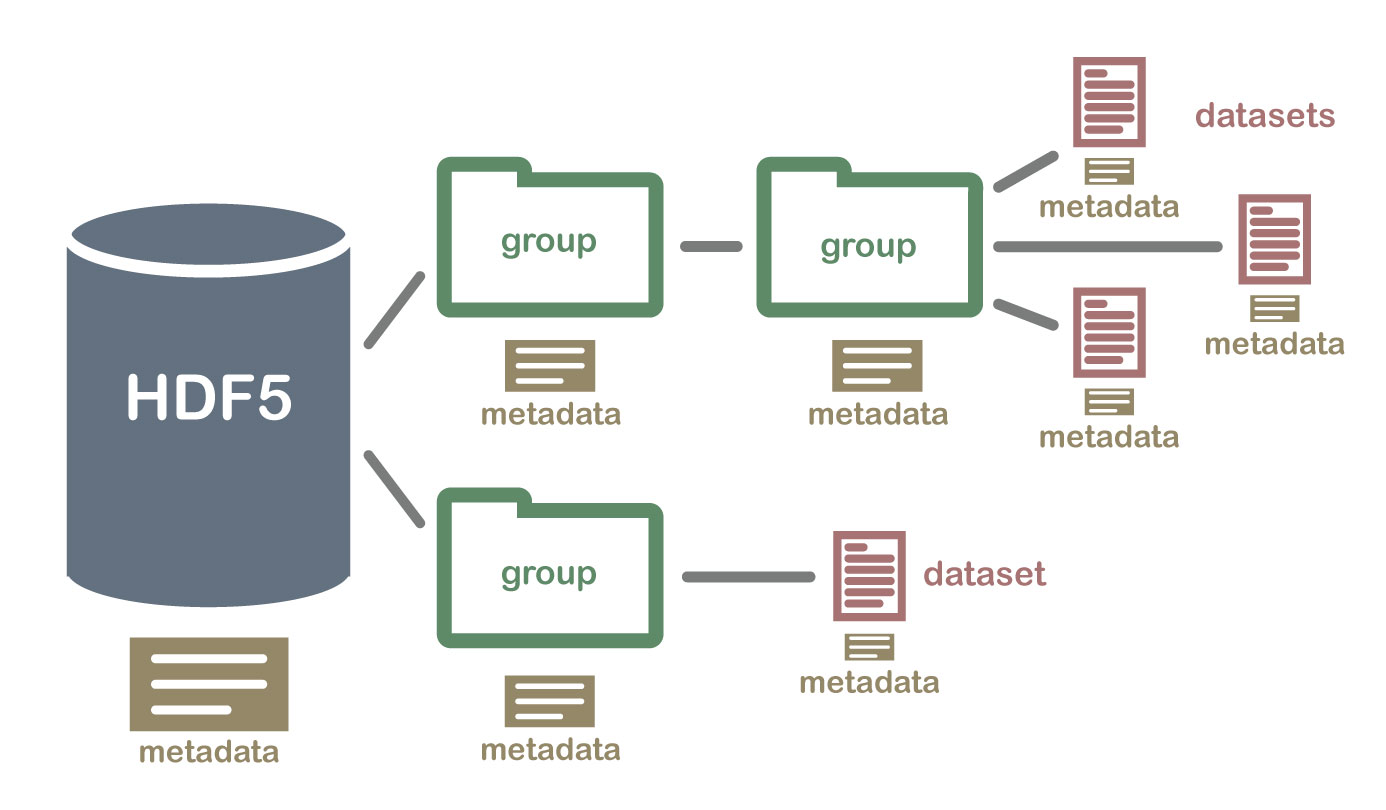
HDF5를 설명하는 다이어그램 (이미지 소스: neondataskills.org)
HDF5 파일은 데이터셋과 그룹이라는 두 종류의 객체를 담기위한 컨테이너입니다. 데이터셋은 배열과 유사한 형태의 데이터 모음(collection)이고, 그룹은 폴더와 유사한 형태의 컨테이너인데 데이터셋과 다른 그룹을 담을 수 있습니다. h5py를 사용할 때 가장 근본적인 것은 이것입니다:
그룹은 사전처럼 작동하고, 데이터셋은 Numpy 배열처럼 작동한다.
h5py를 사용하기 위해 맨 처음 해야할 일은 다음과 같이 새 파일을 생성하는 것입니다:
import h5py
import numpy as np
f = h5py.File('testfile.hdf5', 'w')
파일객체는 시작점입니다. 그리고 이것은 데이터 계층에서 루트 폴더 ‘/’의 위치를 차지합니다. 이것은 몇가지 흥미로운 메소드들을 포함하는데, 그중의 하나는 create_dataset입니다:
dset = f.create_dataset("mydataset", (100,), dtype='i')
만들어진 데이터셋 객체는 배열은 아니지만, HDF5 데이터셋입니다. 마치 Numpy 배열과 같이, 데이터셋은 shape와 data type을 가집니다:
>>> dset.shape
(100,)
>>> dset.dtype
dtype('int32')
데이터셋은 또한 배열처럼 슬라이싱을 지원합니다. 이 방법을 통해서 파일에 있는 데이터셋으로부터 데이터를 읽어들이거나 기록할 수 있습니다:
>>> dset[...] = np.arange(100)
>>> dset[0]
0
>>> dset[10]
10
>>> dset[0:100:10]
array([ 0, 10, 20, 30, 40, 50, 60, 70, 80, 90])
더 알기 위해서는, 파일객체와 데이터셋들을 살펴봅시다.
Groups and hierarchical organization
“HDF”라는 말은 “Hierarchical Data Format”의 의미입니다. HDF5 파일의 모든 객체는 이름을 가지는데 / 구분자를 사용하여 POSIX스타일의 계층 구조로 배열됩니다:
>>> dset.name
u'/mydataset'
이 시스템에서 “폴더”를 그룹이라고 합니다. 우리가 만든 파일 객체는 그 자체로 그룹입니다. 이 경우에는 / 라고 부르는 루트 그룹입니다:
>>> f.name
u'/'
서브그룹 생성은 알맞은 이름이 주어진 create_group을 통해서 수행됩니다:
>>> grp = f.create_group("subgroup")
또한 모든 그룹 객체에는 파일처럼 create_* 메소드도 있습니다:
>>> dset2 = grp.create_dataset("another_dataset", (50,), dtype='f')
>>> dset2.name
u'/subgroup/another_dataset'
그런데 모든 중간 그룹을 수동으로 만들 필요는 없습니다. 전체경로를 한번에 지정하는 것도 잘 작동합니다:
>>> dset3 = f.create_dataset('subgroup2/dataset_three', (10,), dtype='i')
>>> dset3.name
u'/subgroup2/dataset_three'
그룹은 대부분의 파이썬 사전스타일의 인터페이스를 지원합니다. 사전에서 항목을 검색하는 것처럼, 다음과 같은 구문을 사용하여 파일의 객체를 검색할 수 있습니다:
>>> dataset_three = f['subgroup2/dataset_three']
마치 사전처럼, 그룹에 대한 반복은 멤버의 이름을 제공합니다:
>>> for name in f:
... print name
mydataset
subgroup
subgroup2
포함여부를 시험(containership testing)하는 것에서도 이름을 사용합니다:
>>> "mydataset" in f
True
>>> "somethingelse" in f
False
포함여부의 시험에 전체경로 이름을 사용할 수도 있습니다:
>>> "subgroup/another_dataset" in f
True
익숙한 메소드인 get() 뿐만 아니라 keys(), values(), items(), iter() 메소드도 있습니다.
그룹을 반복연산(iterating)하면 직접 연결된 멤버만을 넘겨주므로, 호출가능자(함수)를 인수로 가지는 그룹 메소드인 visit(), visititems()를 사용함으로써 전체 파일을 반복할수 있습니다:
>>> def printname(name):
... print name
>>> f.visit(printname)
mydataset
subgroup
subgroup/another_dataset
subgroup2
subgroup2/dataset_three
Attributes
HDF5의 가장 큰 특징 중 하나는 설명하는 데이터 옆에 메타데이터를 바로 저장할 수 있다는 것입니다. 모든 그룹과 데이터셋은 속성이라는 이름의 데이터 비트를 지원합니다.
메타데이터란 다른 데이터를 기술하거나 다른 데이터에 관련한 정보를 제공하는 데이터의 집합을 말합니다. 예를 들어서 어떤 기상예측 시뮬레이션 모델을 실행해서 예측결과를 얻었는다고 할때, 시뮬레이션 결과는 데이터셋에 저장하고 시뮬레이션 모델를 실행하는데 필요한 파라미터를 메타데이터로써 저장합니다.
속성은 attrs 프록시 객체를 통해 액세스되며 사전객체를 다시 구현합니다:
>>> dset.attrs['temperature'] = 99.5
>>> dset.attrs['temperature']
99.5
>>> 'temperature' in dset.attrs
True
Data exchange between h5py and Pandas
파이썬의 데이터프레임지원 라이브러리인 pandas가 자체적으로 HDF5형식의 파일 입출력을 지원하긴 하지만, pandas 데이터를 추출하여 직접 HDF5 파일로 쓰려면, h5py의 데이터셋과 속성을 이용하여 pandas의 데이터, 인덱스, 컬럼의 값들을 아래 코드와 같이 저장해 주면 됩니다:
def make_index(raw):
index = raw.astype('U')
if index.ndim > 1:
return pd.MultiIndex.from_tuples(index.tolist())
else:
return pd.Index(index)
with h5py.File('data.hdf5', 'w') as file:
dataset = file.create_dataset('dataset', data=df.values)
dataset.attrs['index'] = np.array(df.index.tolist(), dtype='S')
dataset.attrs['columns'] = np.array(df.columns.tolist(), dtype='S')
with h5py.File('data.hdf5') as file:
dataset = file['dataset']
index = make_index(dataset.attrs['index'])
columns = make_index(dataset.attrs['columns'])
df = pd.DataFrame(data=dataset[...], index=index, columns=columns)
코드의 원문은 여기에서 발췌하였습니다.
Hickle - a HDF5-based python pickle replacement
HDF5가 강력한 기능을 제공하기는 하지만, 이것을 사용하기 위해서는 HDF5만의 방식을 조금은 배워야 할 것입니다. HDF5를 배우는 대신에, 익숙한 파이썬의 pickle 인터페이스를 이용하여 HDF5를 사용할 수도 있습니다. Hickle은 HDF5 기반의 Pickle 클론입니다. Hickle은 피클 파일로 직렬화하는 대신 HDF5 파일로 덤프합니다. 즉, pickle(일반 데이터 객체 용)의 “drop-in” 대체품으로 설계되었습니다. Hickle은 빠르며 특히 데이터의 (LZF / GZIP)와 같은 표준 프로토콜로 압축할 수 있습니다.
주의) hickle은 아직 최신버전의 파이썬을 지원하는데 문제가 있는 것으로 보입니다.
Hickle 다운로드 및 설치하기: https://github.com/telegraphic/hickle
Matlab example
HDF5는 matlab에서도 사용할 수 있습니다. HDF5가 공통의 형식이기 때문에, 이를 이용하여 다른 언어로 작성된 프로그램들과 데이터를 상호교환할 수 있을 것입니다.
matlab에서는 hdf5read, hdf5write, hdf5info를 지원합니다.
dset = single(rand(10,10));
hdf5write('myfile2.h5', '/group1/dataset1', dset, '/group1/names', {'a','b','cde'});
hdf5info('myfile2.h5')
hdf5read('myfile2.h5', '/group1/dataset1')
hdf5read('myfile2.h5', '/group1/names')
writemode는 overwrite나 append가 될 수 있습니다.
